- 0
- 국내산라이츄
- 조회 수 7382

아 별을 이모지로 넣어달라고요? 리눅스는 이모지 어떻게 열어요? (이사람)
*배경 설명이 필요하신 분은 https://studyforus.com/koreanraichu/246304
를 읽어보고 오시는 걸 추천드립니다.
0. 공통 기능
1) Database를 가져와서 거기 있는 데이터대로 검색하고 출력합니다. DB는 csv파일로 깃헙에 올라가 있고, 두 개가 있는데 한쪽은 인식하는 시퀀스나 자르는 영역이 A, T, G, C만 들어가는 효소들이 있고 다른 DB는 뭐 알파벳이 아주 개판입니다... W(A or T) S(G or C) 막 이래서 이거는 정규식 처리를 해야 합니다... (대환장파티) 그래서 cutter와 finder는 정규식이 필요 없는 DB를 쓰고, Searcher는 그냥 검색엔진같은 거라서 두 개 부른 다음 합쳐서 씁니다. (자체 구축하느라 개고생함)
2) cutter와 finder의 경우 출력 형식이 일정하게 정해져 있습니다. searcher는 출력은 해 주는데 저장은 안 해줍니다.
3) cutter와 finder에서 FASTA 파일을 지원할 예정입니다. (꺾쇠 하나짜리만) FASTA 파일을 올릴 경우 시퀀스에 대한 정보도 FASTA 파일에서 가져옵니다.
4) (대공사 예정) cutter와 finder에서도 정규식을 이용해 W, S, N같은 괴랄한 알파벳들을 처리할 수 있게 할 예정입니다. ...아니 그럼 함수가 몇 개야...
1. Finder(모든 코드의 시발점)
https://github.com/koreanraichu/CodingPDS/blob/master/python/Restriction%20enzyme%20finder.py
이건 말 그대로
1. DNA 시퀀스를
2. 특정 제한효소가
3-1. 자르는가?
3-2. 자른다면 어디를 얼마나 자르는가?
를 찾는 코드입니다.
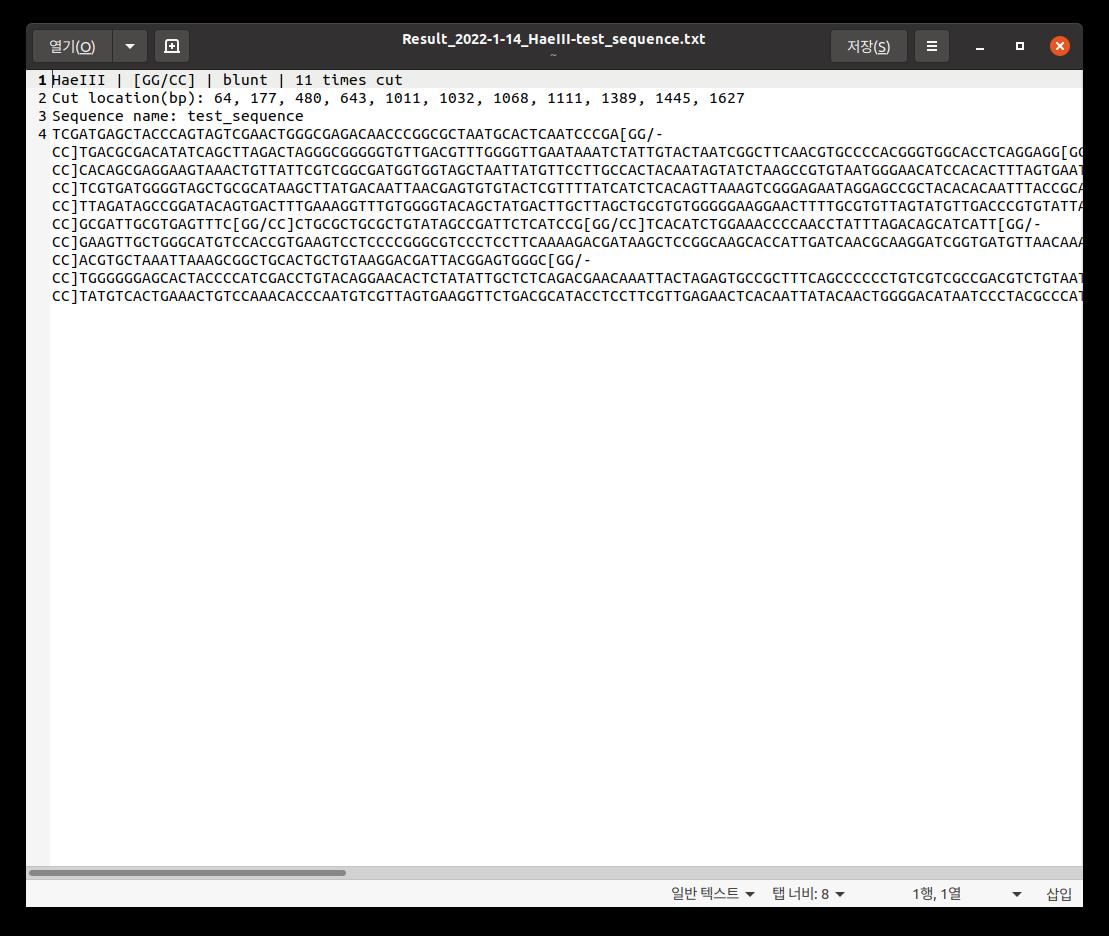
그리고 찾은 결과를 텍스트파일로 출력도 해 줍니다. 근데 효소 이름이... 해삼이요? 효소가 인식하고 자르는 부분은 [시/퀀스]로 표시됩니다. 위 사진에 보면 [GG/CC]로 표시된 부분이 있는데 HaeIII이 인식하고 자르는 부분입니다.
2. Cutter(1차 파생)
https://github.com/koreanraichu/CodingPDS/blob/master/python/Restriction%20enzyme%20cutter.py
이건 Finder랑 좀 개념이 다릅니다. 비슷한 tool로는 NEB cutter가 있는데 거기께 더 좋으니까 걍 그거 쓰세요. 이렇게 tool 홍보를
이 코드는
1. 시퀀스를 입력하면
2. 제한효소 시퀀스를 싹 다 찾아서
3. 얘가 어디를 자르는지를 알려줍니다.
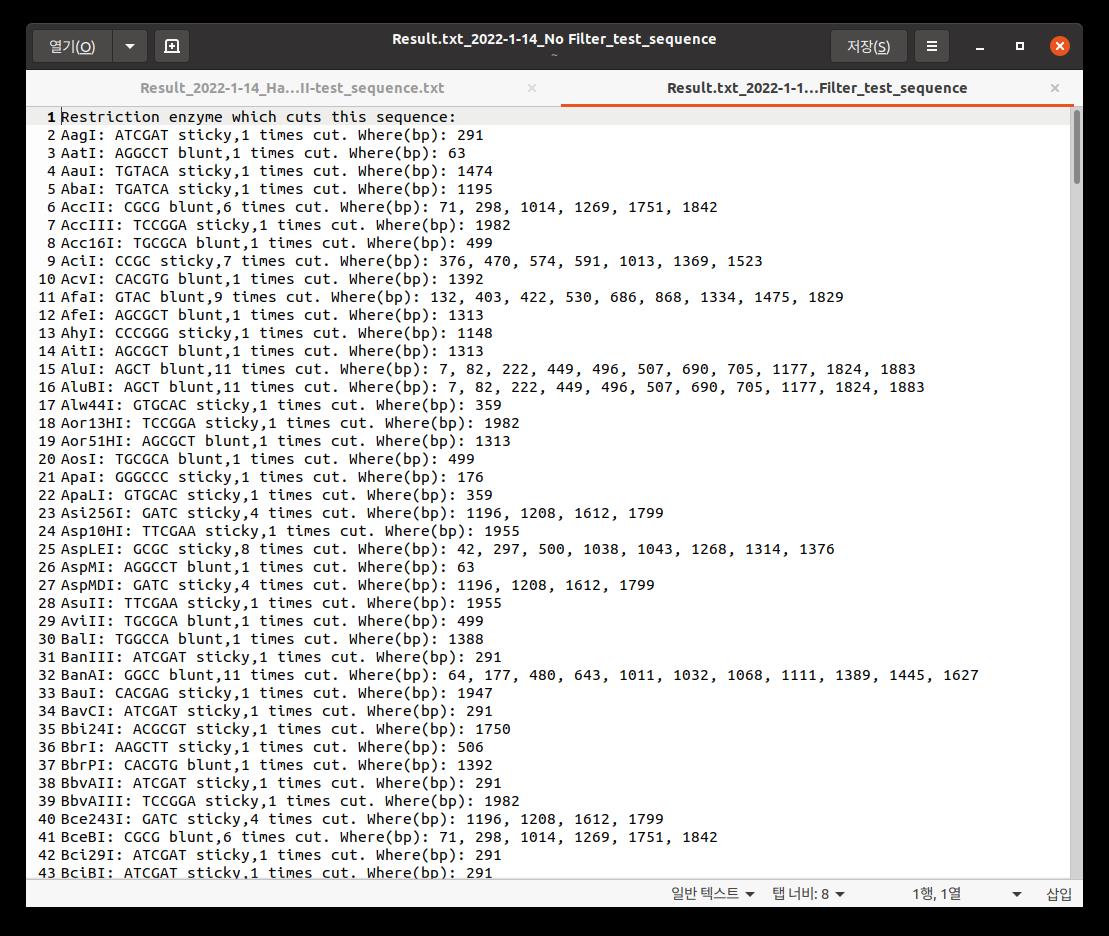
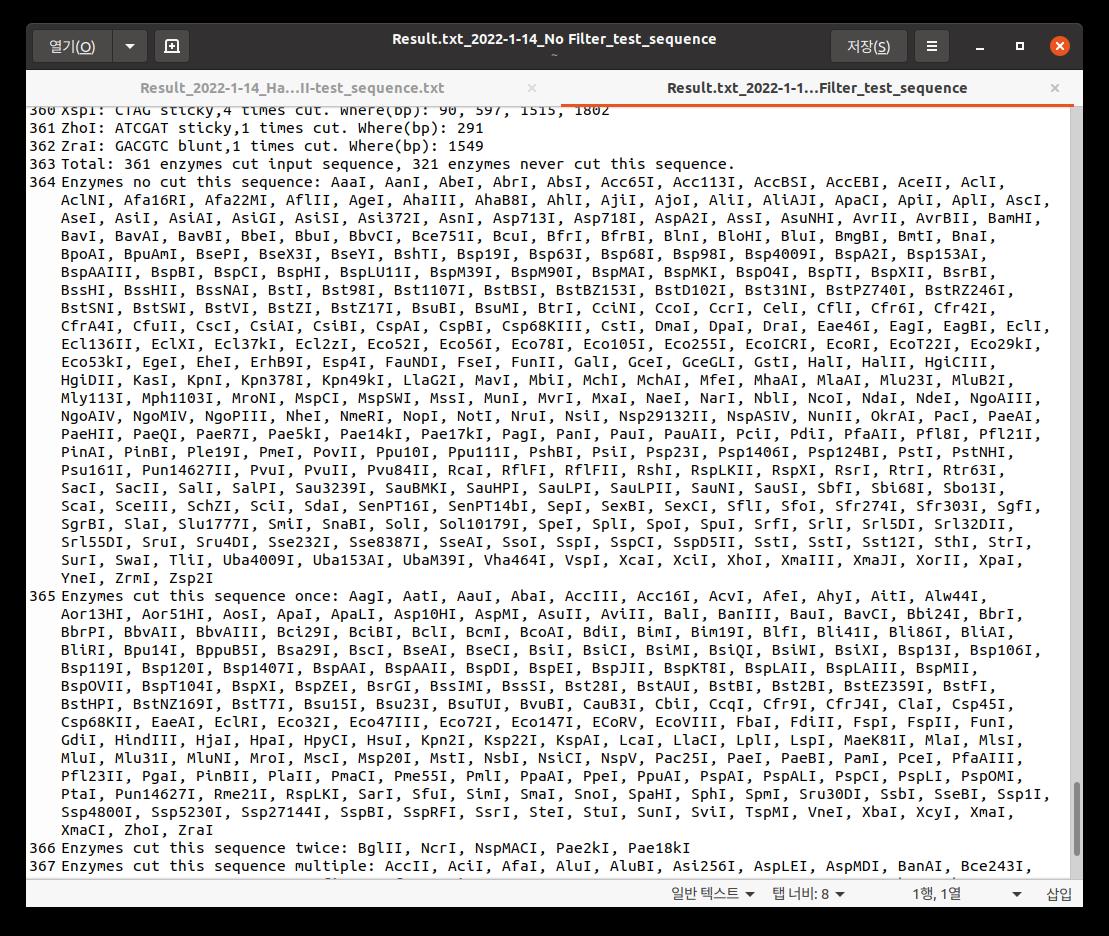
얘는 그래서 DB에 있는 모든 제한효소들의 인식 시퀀스를 검색한 다음 0컷 1컷(한번 자름) 2컷(두번 자름) 멀티컷(3회 이상 자름)으로 나눠서 친절하게 기록까지 해 줍니다.
3. Searcher(마지막 파생)
https://github.com/koreanraichu/CodingPDS/blob/master/python/Restriction%20enzyme%20searcher.py
이 코드는 결과가 따로 저장되지는 않고
1. 제한효소의 이름으로 검색해서 똑같은 시퀀스를 인식하는 효소를 찾아주거나
2. 제한효소의 인식 시퀀스를 알면 그걸로 검색하거나
3. 아 그 뭐더라같은 느낌으로 이름이 생각 안 날 때 효소 머릿글자로 검색하거나(급하면 그럴 때 있음) 그렇습니다.

(그 뭐더라 D로 시작하는 제한효소 있는데)
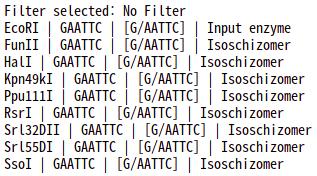
(EcoRI으로 검색한 결과)
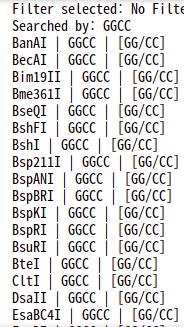
(인식 시퀀스 GGCC로 검색한 결과)



 허니버터라이츄 | 2017.10.30
허니버터라이츄 | 2017.10.30